前 言
单次实验即可获得上万个细胞的基因表达数据(大老远都觉得香),直接把分辨率从组织水平干到了单个细胞水平(当然,最后说事大多数还是基于细胞cluster或者sub cluster)。单细胞的生信分析会对数据进行可视化。基于tSNE或者UMAP等算法的细胞聚类分群的可视化展示是基于数学逻辑的,尚无生物学意义。当前主流的做法之一是通过marker基因将细胞亚群进行类型定义。这里就需要用到细胞类型标记基因数据库,这是单细胞分析最为耗时的关键步骤。
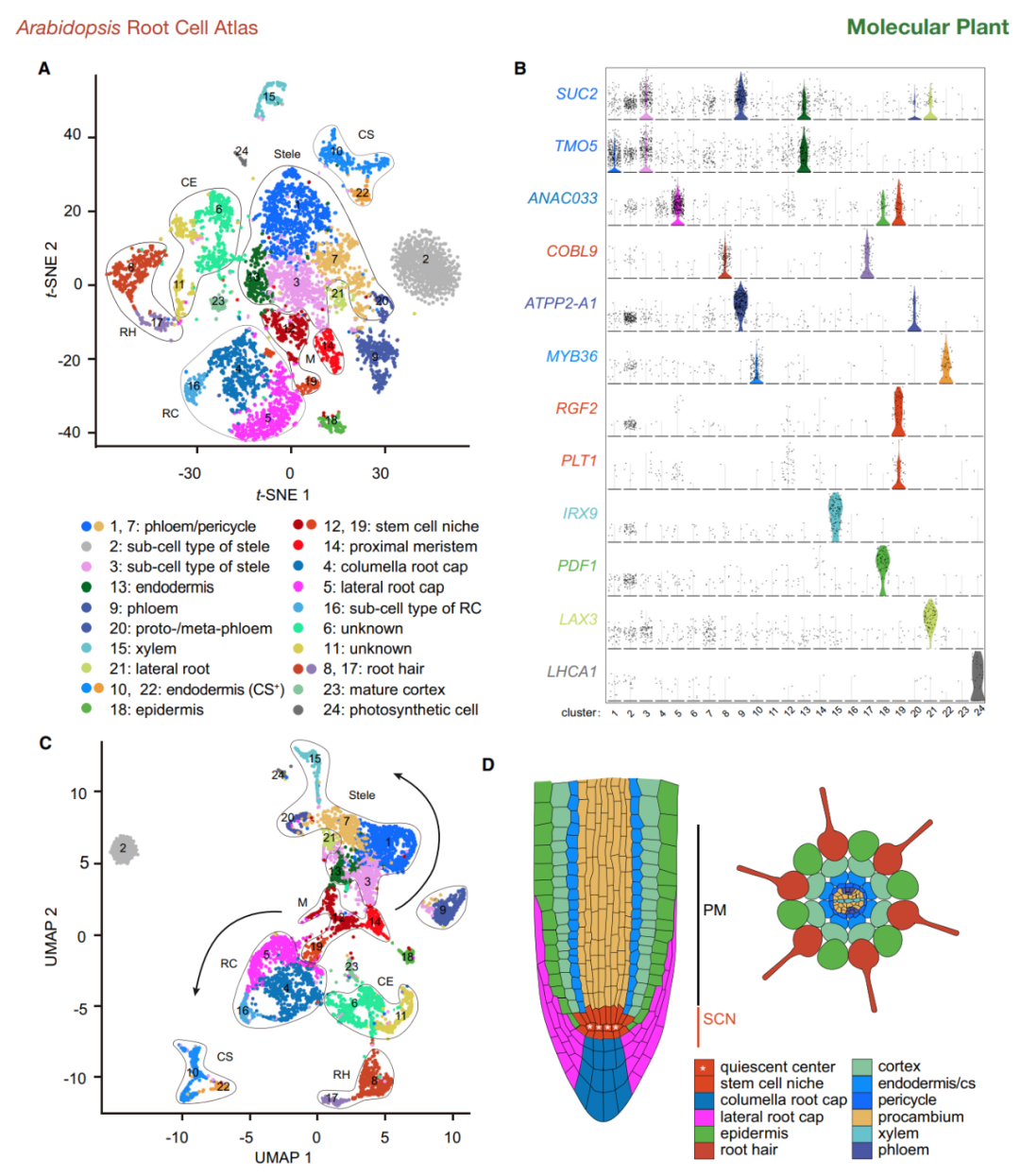
下图为发表的拟南芥根的细胞分群及类型定义[1],小编觉得很赏心悦目。
数据库链接:http://ibi.zju.edu.cn/plantscrnadb/index.php
论文发表于:Molecular Plant(2021)[2]
PlantscRNAdb是浙江大学樊龙江教授团队开发的一个植物单细胞RNA分析数据库。新的3.0版本收录了15个物种(拟南芥、水稻、番茄、玉米、野草莓、杨树、烟草、小柠檬、油菜、木薯、苜蓿、荆芥、陆地棉、大豆、白蝴蝶兰),38个组织,451种细胞类型,114770个marker基因。

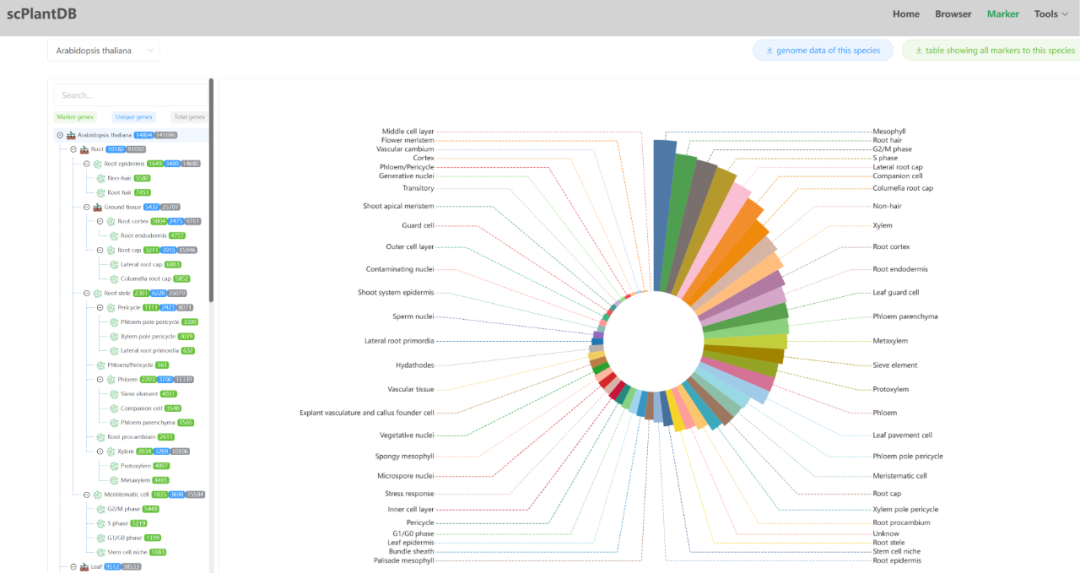
数据库链接:https://biobigdata.nju.edu.cn/scplantdb/home
论文发表于:Nucleic Acids Research(2023)[3]
scPlantDB是南京大学生命科学学院陈迪俊教授课题组构建的植物单细胞综合数据库平台。该数据库整合了来自17个植物物种(拟南芥、水稻、玉米、棉花、番茄、84K杨树、苜蓿、小麦、烟草、木棉树、大白菜、木薯、草莓、大豆、新疆杨、比克氏棉、长春花)的67套高质量单细胞图谱,涵盖约250万个细胞,包含259种细胞类型和近23万个标记基因。


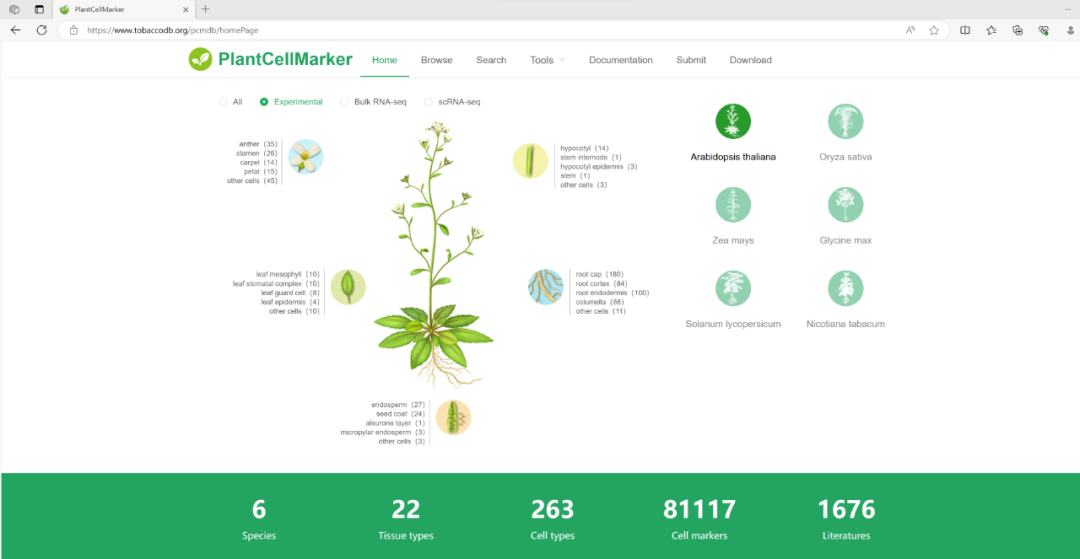
03、PCMDB
数据库链接:https://www.tobaccodb.org/pcmdb/homePage
论文发表于:Nucleic Acids Research(2022)[4]
PCMDB(Plant Cell Marker Database)是郑州烟草研究院国家烟草基因研究中心曹培健团队开发建立。PCMDB基于三个数据来源(实验验证、普通转录组数据、单细胞转录组数据),挖掘了拟南芥、水稻、玉米、大豆、番茄、烟草等6个模式植物的81117个植物细胞标记基因,覆盖了22个组织的263种细胞类型。

数据库链接:http://jinlab.hzau.edu.cn/PsctH/
论文发表于:Plant Biotechnology Journal (2021)[5]
Plant Single Cell Hub(PsctH)是由华中农业大学作物遗传改良全国重点实验室开发的植物单细胞综合数据库,主要包含5个物种(拟南芥、棉花、水稻、番茄和玉米)的10个植物组织的52种细胞类型的104个细胞标记基因,PsctH中的所有标记基因必须通过RNA原位杂交或GFP报告基因的表达来证明。
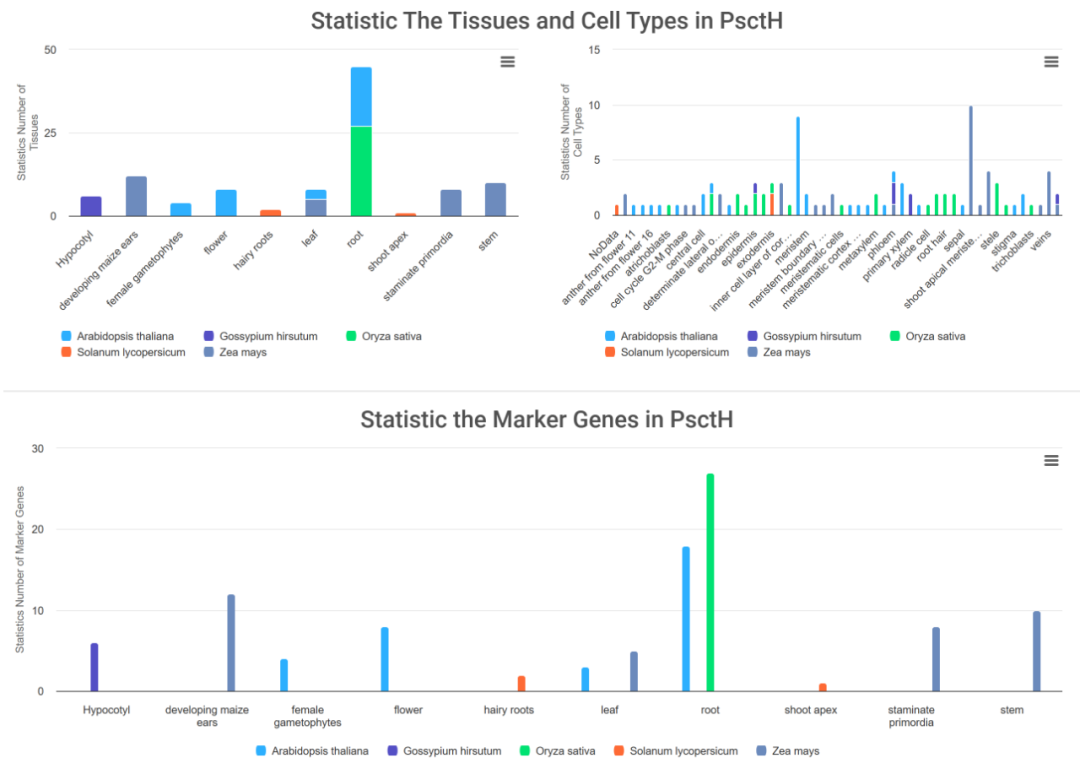
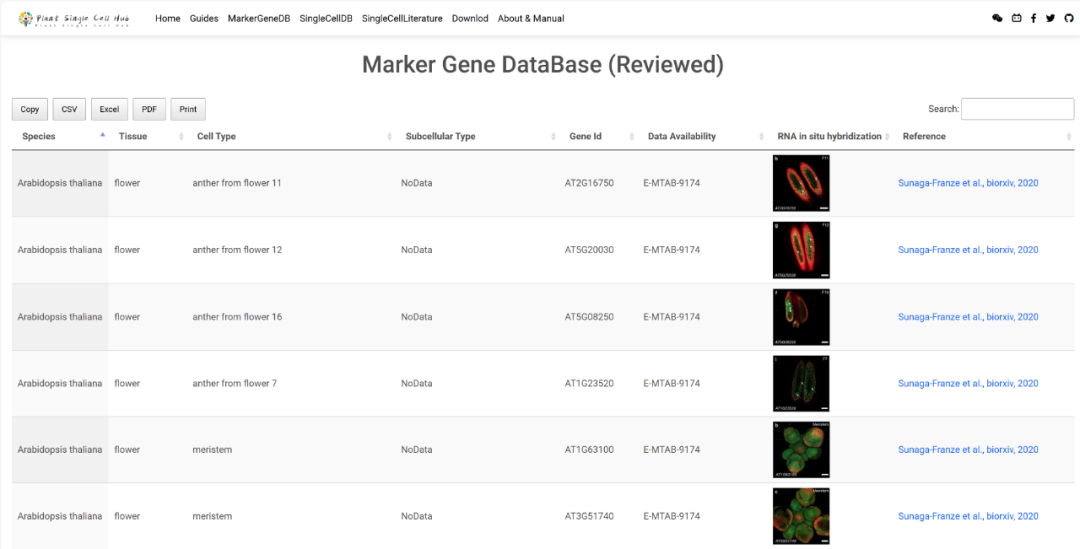
05、RCAR
数据库链接:http://www.elabcaas.cn/rcar/index.html
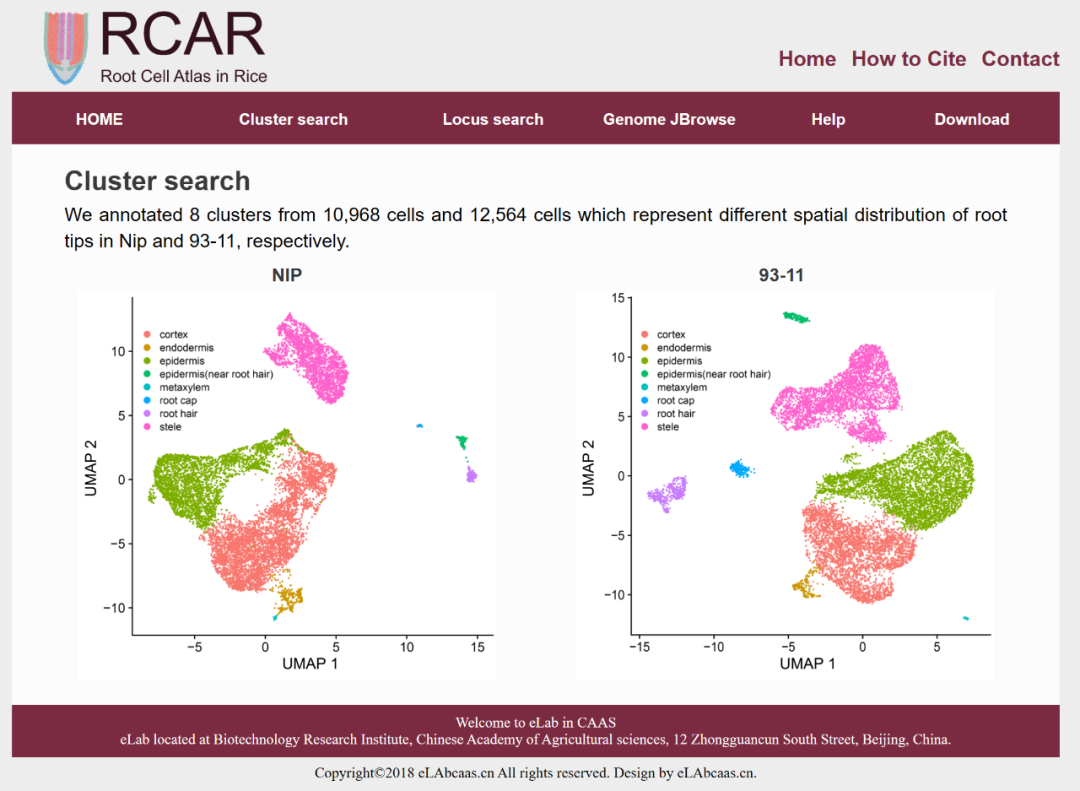
RCAR(Root Cell Atlas in Rice)是中国农业科学院生物技术研究所谷晓峰教授课题组开发的两个水稻栽培品种(粳稻NIP和籼稻93-11)根尖组织单细胞转录图谱数据库。通过各个模块,可以查询各细胞类型或亚群的基因表达,也可以查询基因在不同类型细胞或亚群的表达。

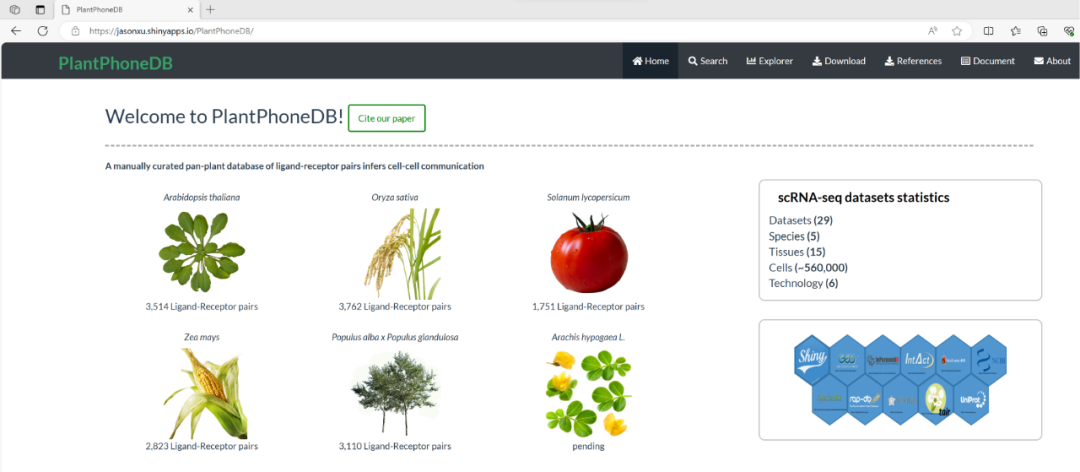
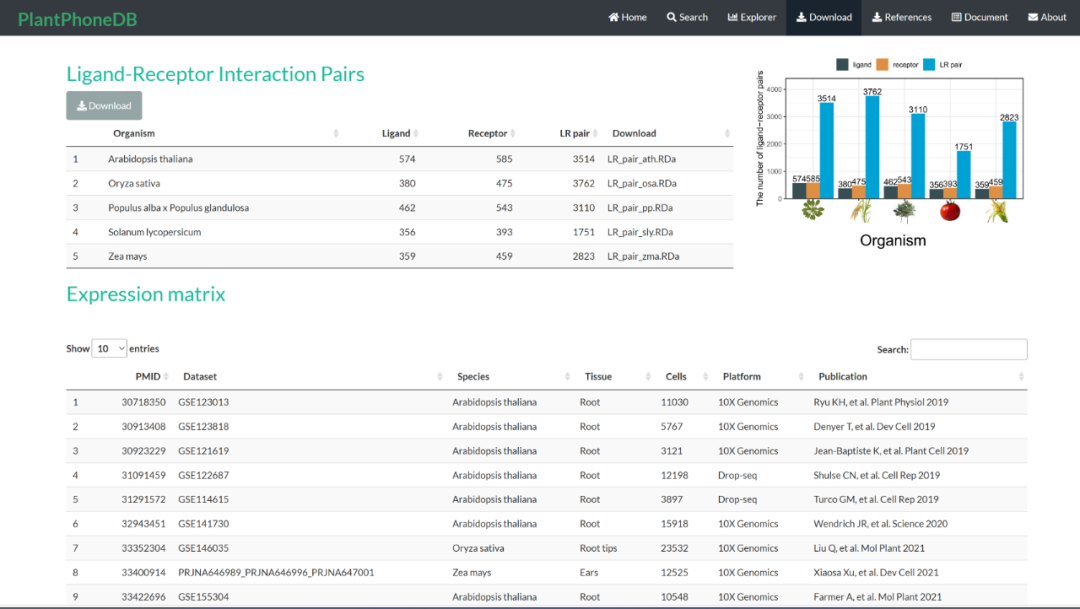
数据库链接:https://jasonxu.shinyapps.io/PlantPhoneDB/
论文发表于:Plant Biotechnology Journal (2022)[6]
PlantPhoneDB是一个手工整理的可用于细胞通讯分析的植物配体-受体数据库。目前数据库收录了拟南芥、水稻、番茄、玉米和杨树五个物种的大量高可信度配体-受体数据。从页面看,花生的数据正在建设中。该数据库可搜索指定配体或受体的详细信息,浏览数据库已有的单细胞数据集对应的分析结果并下载。
小 结
从整理结果看,目前植物领域的细胞标记基因数据库均为国人建设开发,小编在此致敬。本次分享就到这里,如有其他数据库遗漏,还请后台留言;如有新的数据库,小编会及时更新,敬请留意。
1. Zhang, T.Q., et al., A Single-Cell RNA Sequencing Profiles the Developmental Landscape of Arabidopsis Root. Mol Plant, 2019. 12(5): p. 648-660.
2. Chen, H., et al., PlantscRNAdb: A database for plant single-cell RNA analysis. Mol Plant, 2021. 14(6): p. 855-857.
3. He, Z., et al., scPlantDB: a comprehensive database for exploring cell types and markers of plant cell atlases. Nucleic Acids Res, 2023.
4. Jin, J., et al., PCMDB: a curated and comprehensive resource of plant cell markers. Nucleic Acids Res, 2022. 50(D1): p. D1448-D1455.
5. Xu, Z., et al., Plant Single Cell Transcriptome Hub (PsctH): an integrated online tool to explore the plant single-cell transcriptome landscape. Plant Biotechnol J, 2022. 20(1): p. 10-12.
6. Xu, C., et al., PlantPhoneDB: A manually curated pan-plant database of ligand-receptor pairs infers cell–cell communication. Plant Biotechnol J, 2022. 20(11): p. 2123-2134.
原文地址:https://blog.csdn.net/SHANGHAILINGEN/article/details/134733514
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。
如若转载,请注明出处:http://www.7code.cn/show_19729.html
如若内容造成侵权/违法违规/事实不符,请联系代码007邮箱:suwngjj01@126.com进行投诉反馈,一经查实,立即删除!