本文介绍: 为了解决特征特征数量级差异过大,导致的模型过拟合问题,有一种方法就是对每个特征进行缩放,使其大致处于同一范围。核SVM常用的缩放方法是将所有的特征缩放到0和1之间。
为了解决特征特征数量级差异过大,导致的模型过拟合问题,有一种方法就是对每个特征进行缩放,使其大致处于同一范围。核SVM常用的缩放方法是将所有的特征缩放到0和1之间。
“人工”处理方法:
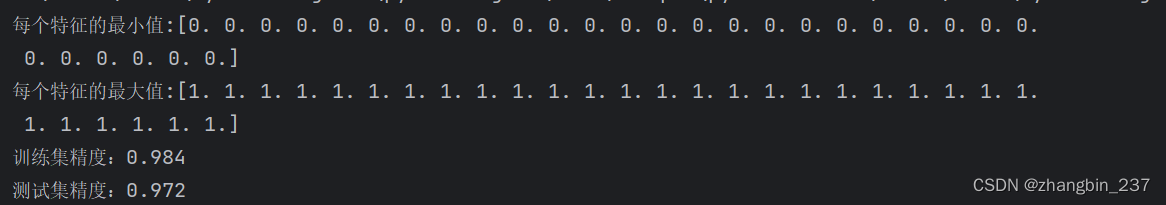
可以看到,最终的结果上训练集和测试集的精度都非常好,但还没有接近100%的精度,可能存在欠拟合,后续可以通过调整C参数来继续优化。
声明:本站所有文章,如无特殊说明或标注,均为本站原创发布。任何个人或组织,在未征得本站同意时,禁止复制、盗用、采集、发布本站内容到任何网站、书籍等各类媒体平台。如若本站内容侵犯了原著者的合法权益,可联系我们进行处理。