赛题介绍
中枢神经系统类疾病长期以来存在着重要的临床未满足需求。据统计,在当前人口老龄化趋势下,阿兹海默(AD)、帕金森病(PD)等神经退行性疾病和脑癌、中风等疾病的治疗费用将达到数万亿美元,由此产生巨额的医疗保健支出。因此,获得高质量、有效的中枢神经系统药物在当前被普遍视为具有重要的科学和商业价值。
随着AI for science的兴起,AI可以在大规模化合物库中筛选具有潜在治疗作用的药物,基于计算机模拟的方法预测药物的药理活性、毒性和药代动力学特性。通过这种方式,AI可以筛选出最有前景的药物候选者,并优化其化学结构,以提高安全性和有效性。
本次学习赛将提供一批分子信息数据,选手需要以AI方法对分子是否可作为CNS(Central Nervous System)药物进行检测,实现AI助力下的CNS药物筛选和研发工作。
原理简介
中枢神经系统(CNS)包括大脑和脊髓,负责处理和调控身体的各种功能。非中枢神经系统(non-CNS)则包括神经元以外的组织,如内分泌系统和免疫系统等。
由于血脑屏障及其上各类转运体的存在,中枢神经系统用药通常需要满足一些特定的特征。药物化学家会根据特定的指标来判断一个药物是否具有成为中枢神经系统药物的潜力,如分子量和拓扑极性表面积(TPSA)不能过大,N原子和极性H原子数量不能过多,溶剂可及表面需在特定范围内等。这些特征为药物发现和优化阶段中判断分子作为CNS药物的潜力提供了启示。
SMILES 分子表达式中包含了丰富的信息。我们可以利用 SMILES 表达式构建 CNS 药物预测的机器学习模型,并可帮助研究人员深入理解中枢神经系统(CNS)药物和非中枢神经系统(non-CNS)口服药物在众多理化性质方面的差异。这将有助于加速 CNS 药物的筛选和研发过程。
赛题教程
请点击下方链接进入本次学习赛的课程主页,本赛题的一系列教程学习资料会陆续上传,请大家关注。
教程链接:https://nb.bohrium.dp.tech/courses/detail/2718054507
赛题理解及科学知识补充
背景知识
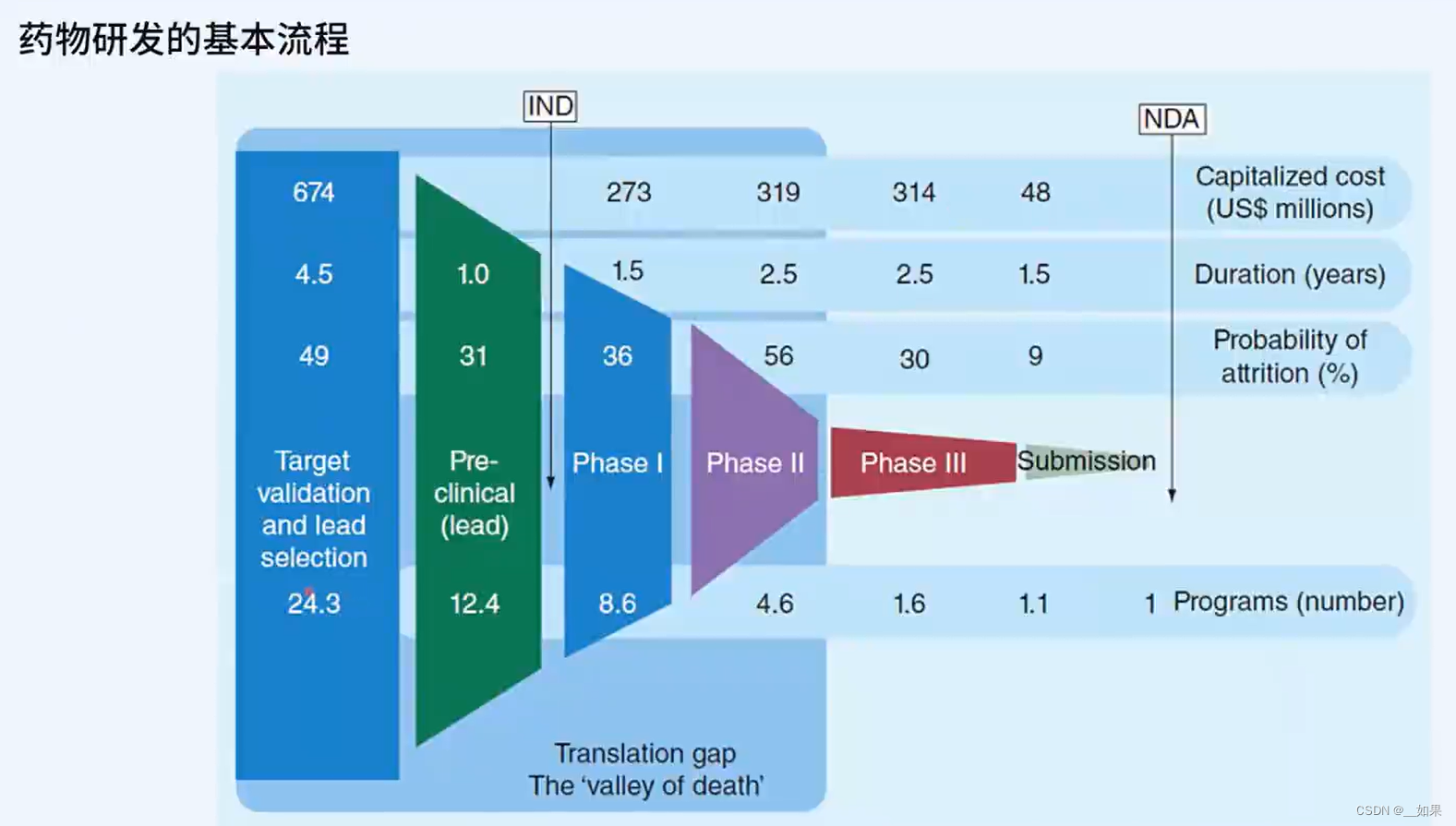
研发一款成功上市的药物需要大约10年的时间,花费10亿美金,平均研发25款药物只有1款能成功提交到药监局,所以迫切希望AI能辅助药物的设计
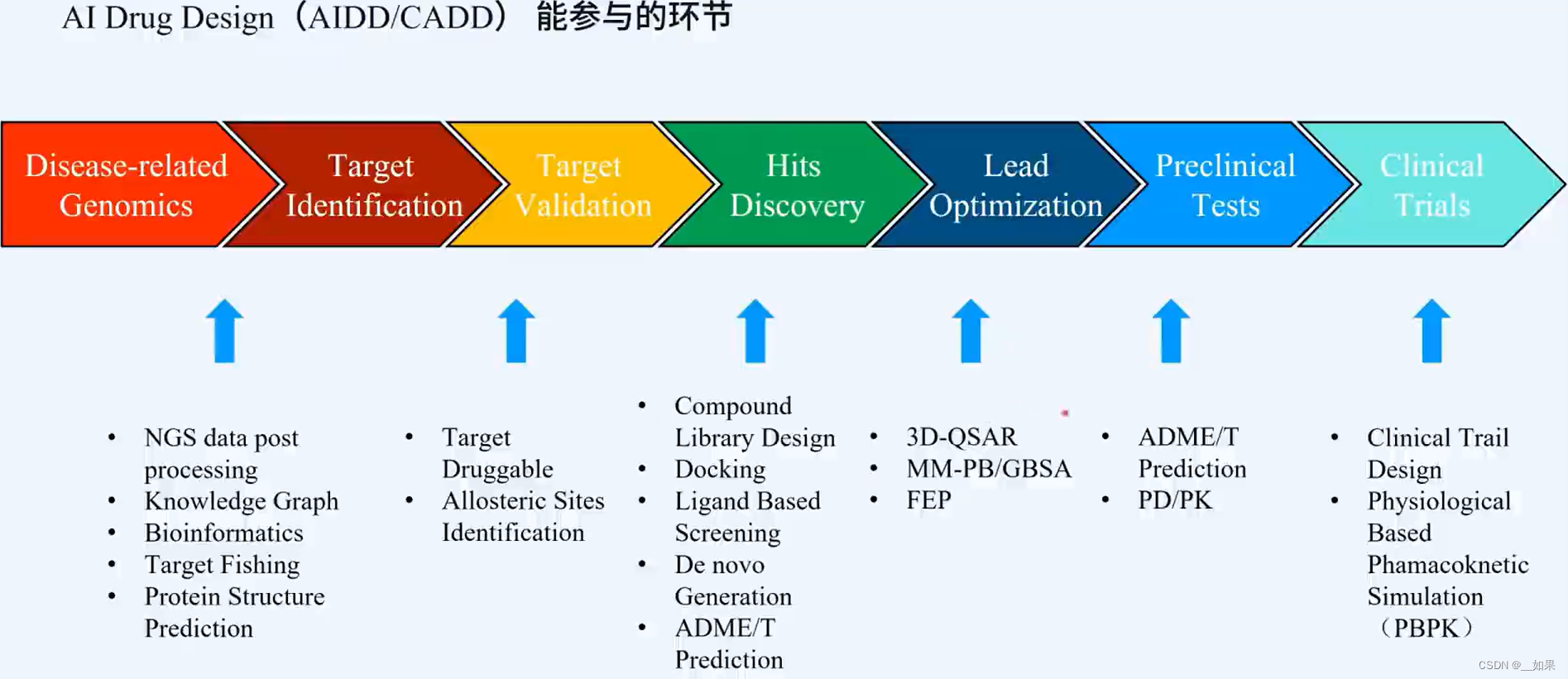
靶点:导致疾病发生的问题的根源,通常是生物体内某些蛋白质出现了功能的失活或失调
对于AD阿茨海默这类疾病,大部分靶点分布在脑部、脊椎等中枢神经系统
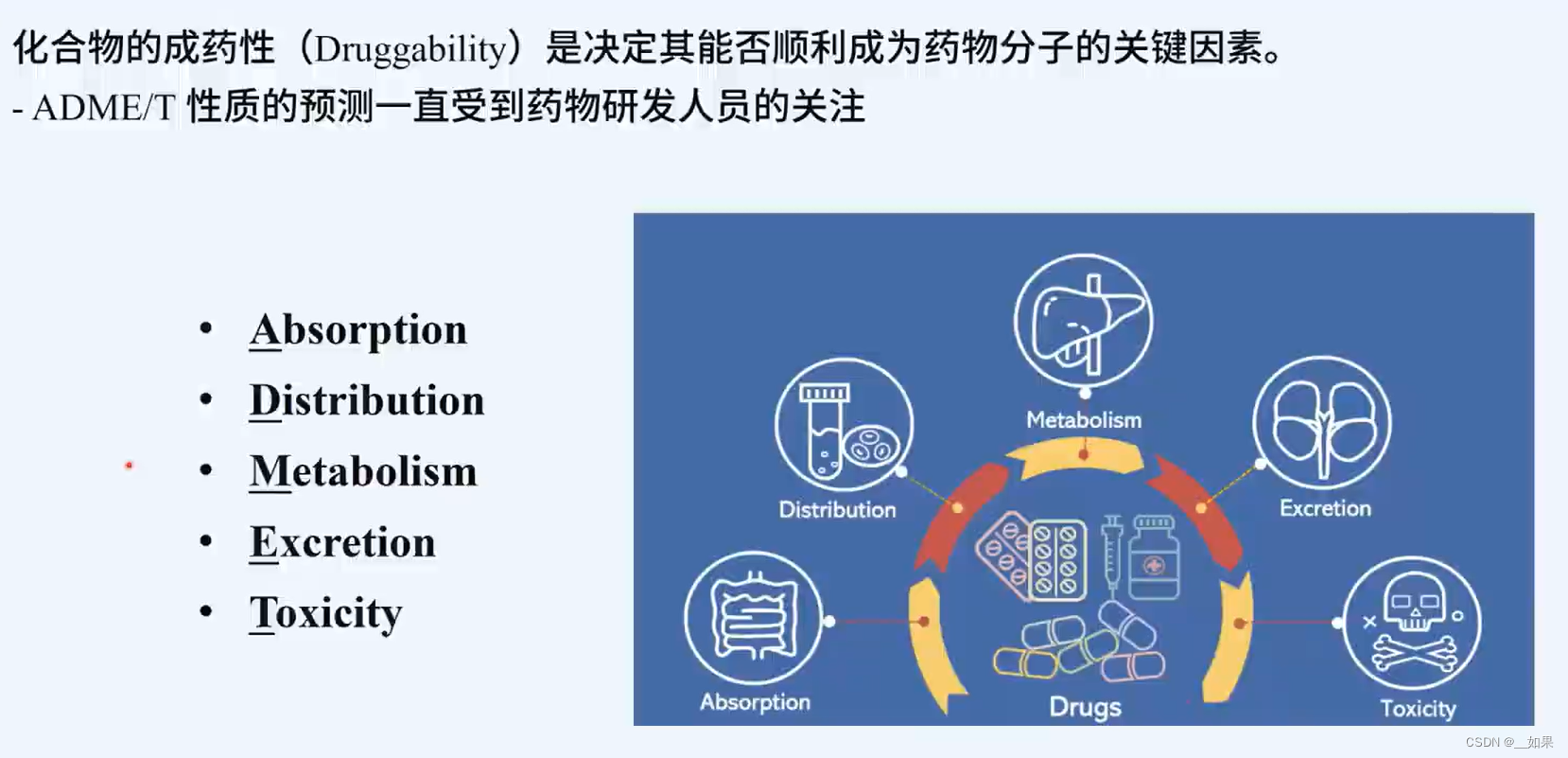
吸收、分布、代谢、排泄、毒性
本次赛题研究的血脑屏障BBB对应着distribution,也就是药物能否顺利的送入中枢神经系统,这是药物能否成为CNS药物的关键

QSAR


QSAR模型的建立需要分子的结构和分子的某些性质,并没有加入任何和这个靶点相互作用的信息
分子结构的表示

赛题背后的逻辑是:比较不同化合物,根据这些化合物的相似性去判断新的化合物与哪些类型的化合物性质相似

对于人来说,系统命名法等方法方便人们去理解,但是对于计算机就不是这回事了


分子特征的表示


常见的定量分子描述符

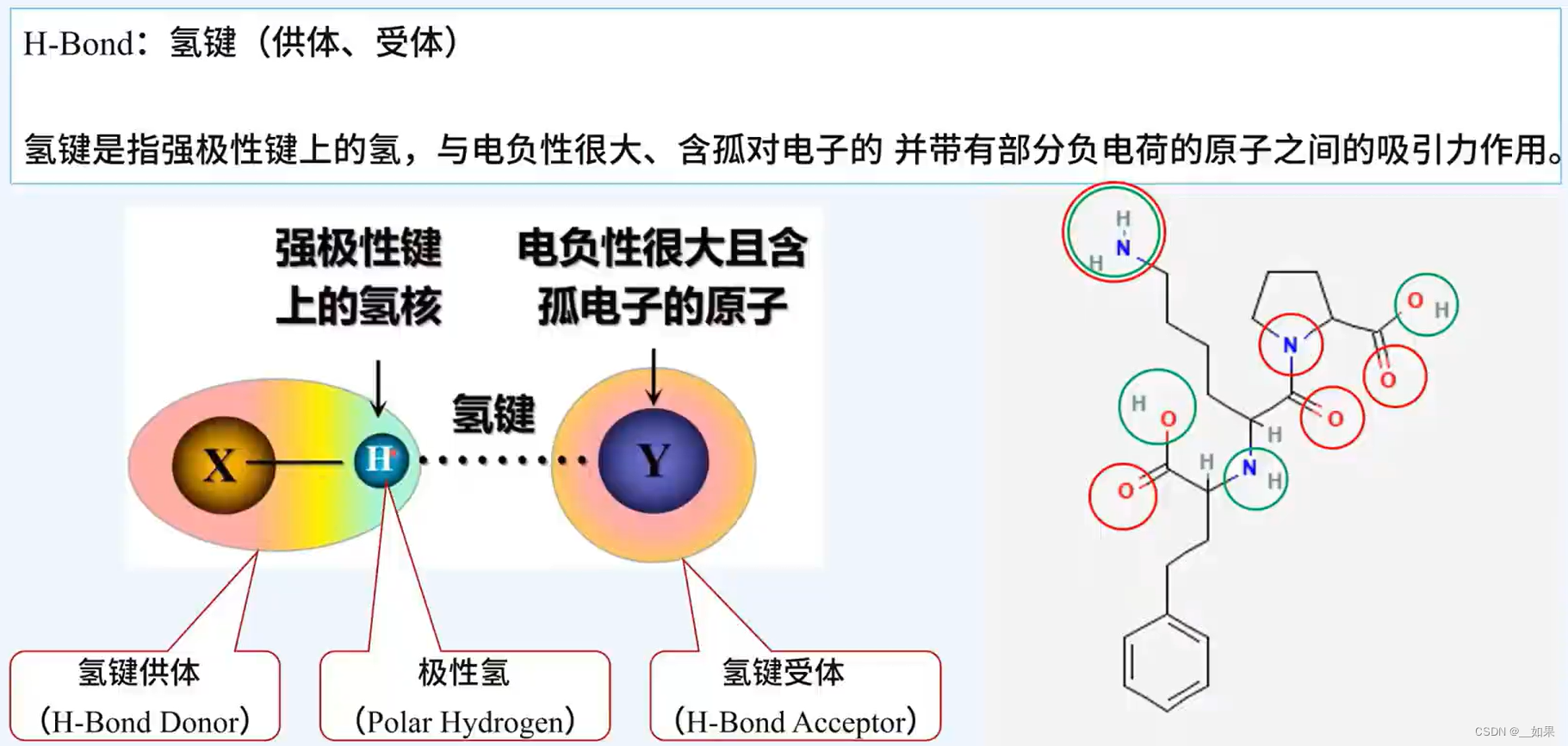
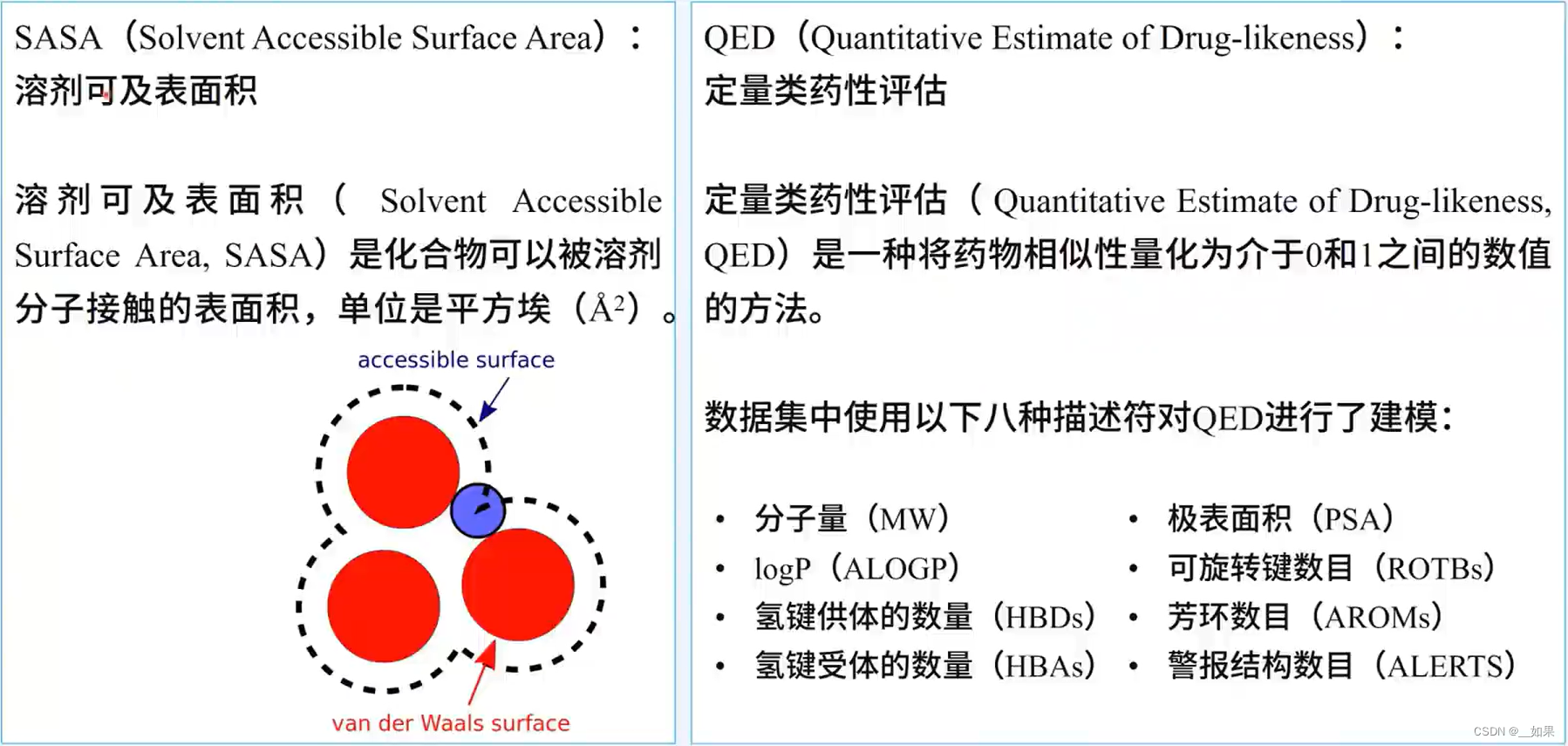
常见的定性分子描述符
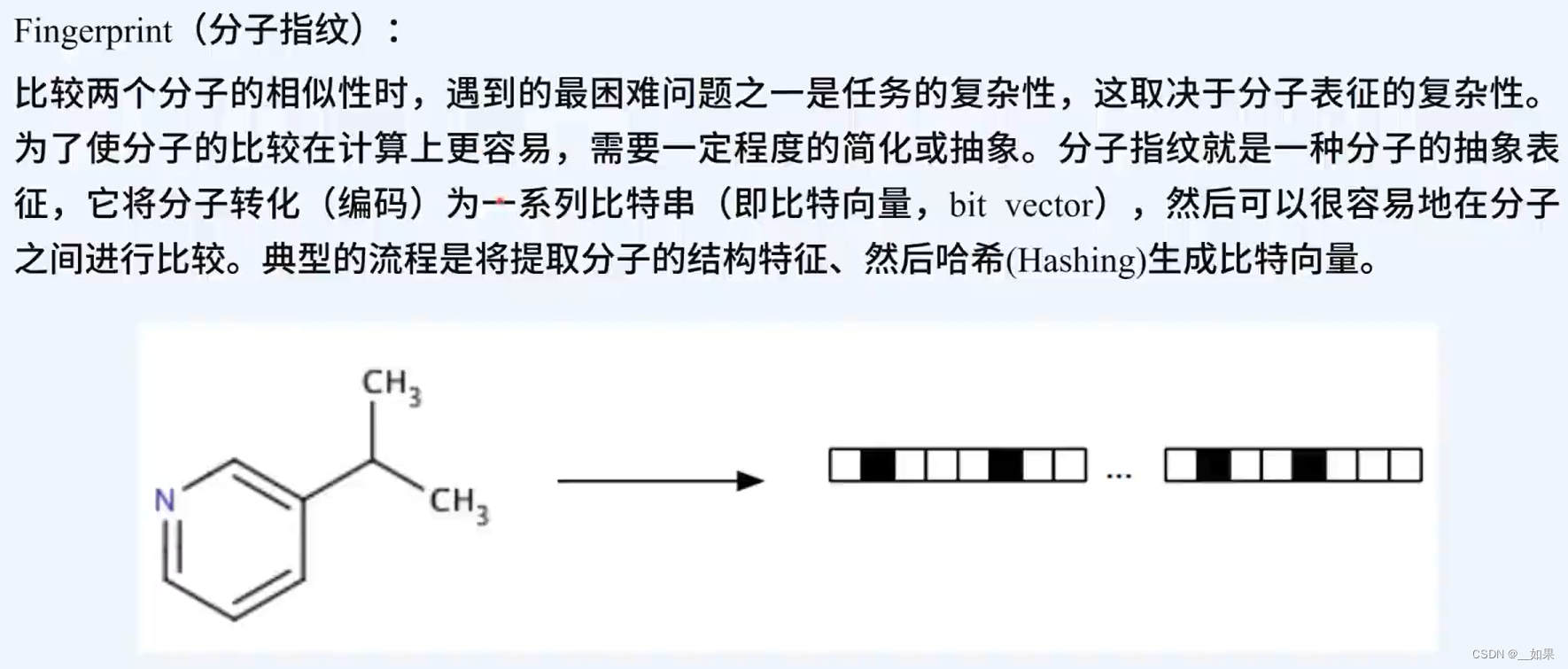
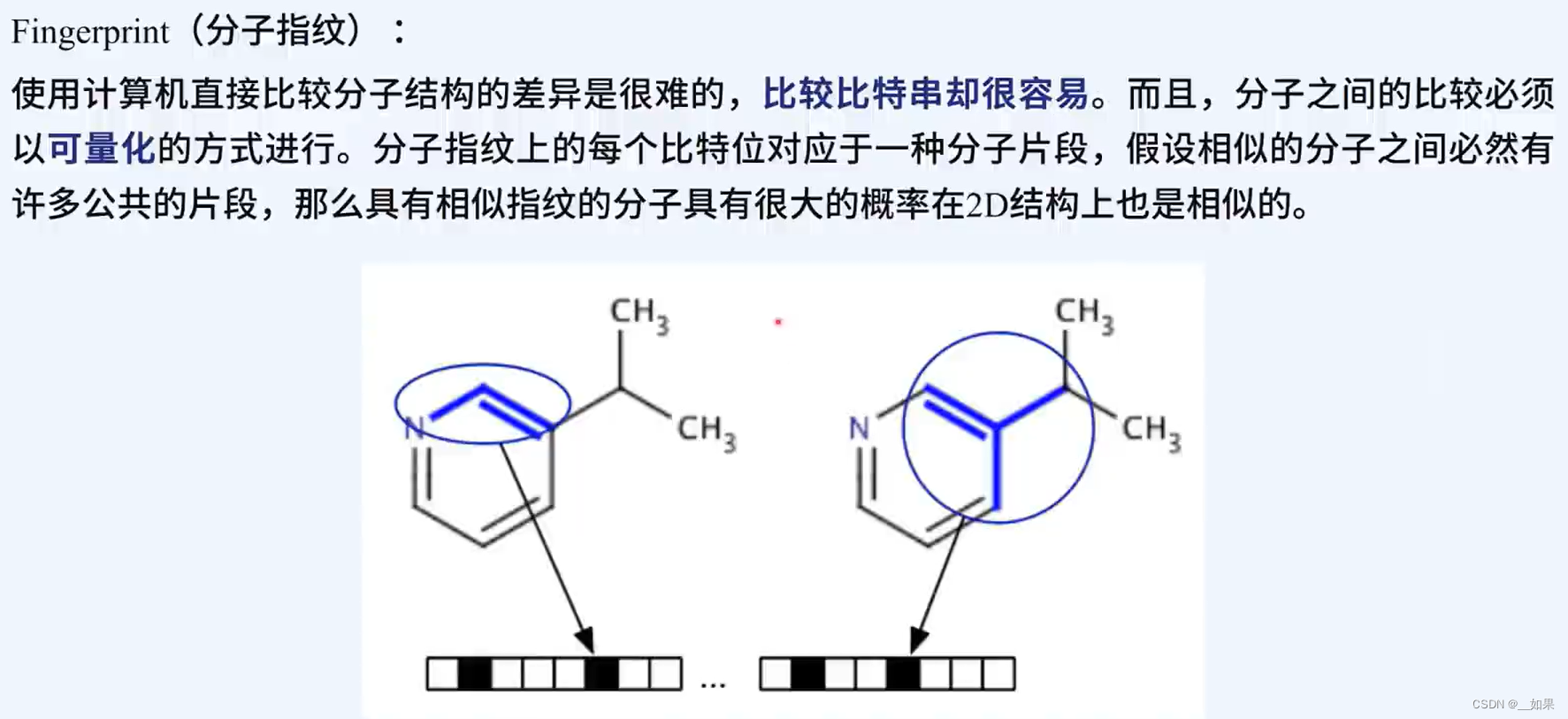

ECFP分子指纹计算流程



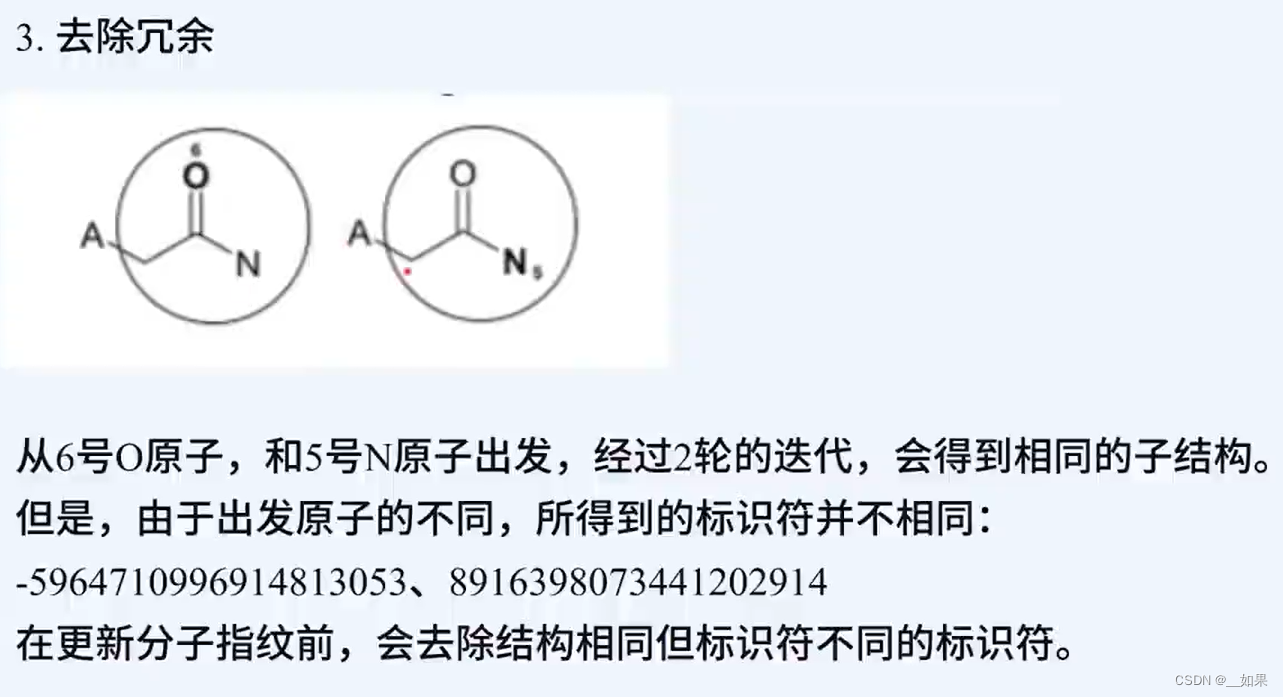

官方baseline
https://nb.bohrium.dp.tech/detail/1151401039
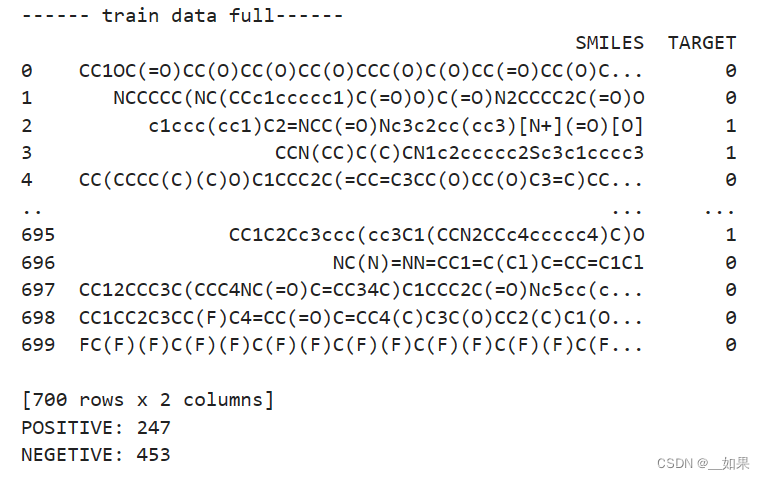
train_data包含两列,一列是SMILES一列是TARGET
TARGET的0代表阴性数据(400多条),1代表阳性数据(200多条)
阳性数据代表这个分子是可以穿过血脑屏障的,有可能成为潜在的治疗CNC的药物




最后使用一些极端随机数、lightgbm、支持向量机等方法验证
baseline
# 需要挂载 bohr CNS 数据集
DIR_PATH = '/bohr/ai4scup-cns-5zkz/v3/'
!pip install lightgbm numpy pandas rdkit scikit-learn
import lightgbm as lgb
import numpy as np
import pandas as pd
from rdkit import Chem # 导入RDKit中的Chem模块,用于分子对象转换
# 从RDKit中导入GetMorganFingerprintAsBitVect函数,用于生成分子指纹(位向量转换)
from rdkit.Chem.rdMolDescriptors import GetMorganFingerprintAsBitVect
from sklearn.model_selection import train_test_split # 从scikit-learn中导入train_test_split函数,用于拆分数据集
from sklearn.metrics import fbeta_score # 从scikit-learn中导入fbeta_score函数,用于 F2 Score 计算
raw_data = pd.read_csv(f"{DIR_PATH}/mol_train.csv")
test_data = pd.read_csv(f"{DIR_PATH}/mol_test.csv")
# 拆分训练数据为训练集与验证集,验证集占比 20%,设定固定随机种子
train_data, valid_data = train_test_split(
raw_data, test_size=0.2, random_state=hash("Datawhale") % 2023
)
def smile2fingerprint(smile: str):
"""将 SMILE 分子式表示为指纹数据
参数:
smile (string): SMILE 分子式
返回:
fp (Explict BitVect): 分子式的 Morgan 指纹位向量
"""
molecular = Chem.MolFromSmiles(smile) # 将字符串转换为分子式对象
finger_print = GetMorganFingerprintAsBitVect(molecular, 5, nBits=1024) # 获得分子式的 Morgan 指纹位向量
return finger_print
# 批量将位向量转换为特征矩阵,形状为 (n, 1024),n 代表数据个数,1024 在位向量转换时指定
train_X = np.array([smile2fingerprint(smile) for smile in train_data["SMILES"]])
valid_X = np.array([smile2fingerprint(smile) for smile in valid_data["SMILES"]])
test_X = np.array([smile2fingerprint(smile) for smile in test_data["SMILES"]])
# 将数据特征矩阵转换为 LightGBM 指定格式,(特征向量,对应标签)
lgb_train = lgb.Dataset(train_X, label=train_data["TARGET"])
lgb_valid = lgb.Dataset(valid_X, label=valid_data["TARGET"])
# 设定 LightGBM 训练参,查阅参数意义:https://lightgbm.readthedocs.io/en/latest/Parameters.html
lgb_params = {
"objective": "binary", # 指定任务类别为二分类
"seed": hash("Datawhale") % 2023, # 设定随机种子
"verbose": -1, # 禁用输出(可选)
}
# 训练模型,参数依次为:导入模型设定参数、导入训练集、设定模型迭代次数(100)、导入验证集
model = lgb.train(lgb_params, lgb_train, num_boost_round=100, valid_sets=lgb_valid)
threshold = 0.5 # 模型输出的是类别概率,设定概率的判断阙值
# 用验证集进行模型预测(选择训练中最好的一次)
valid_pred = model.predict(valid_X, num_iteration=model.best_iteration)
# 生成预测标签结果,如果概率大于阈值则为 1,否则为 0
valid_result = [1 if x > threshold else 0 for x in valid_pred]
# 计算验证集 F2 Score 分数
valid_score = fbeta_score(valid_data["TARGET"], valid_result, beta=2)
print(f"Valid Score: {valid_score}")
# 预测测试集数据并获得预测结果
pred = model.predict(test_X, num_iteration=model.best_iteration)
result = [1 if x > threshold else 0 for x in pred]
submission = pd.DataFrame()
submission["SMILES"] = test_data["SMILES"]
submission["TARGET"] = result
submission.to_csv("./submission.csv", index=False)把finger_print = GetMorganFingerprintAsBitVect(molecular, 5, nBits=1024)中的5改成2,上了一点点分
Chem模块用于将SMILES字符串转化成机器可以识别的数据
这里的finger_print = GetMorganFingerprintAsBitVect(molecular, 5, nBits=1024)其实就对应着科学知识补充中的ECFP指纹——环形指纹
特征工程与可视化
QSAR建模
1.构建合理的分子表征,将分子结构转化为计算机可读的数值表示;
2.选择适合分子表征的机图学习模型,并使用已有的分子-性质数据训练模型;
3.使用训练好的机器学习模型,对未测定性质的分子进行性质预测

1D-QSAR:分子量。可以使用线性回支持向量机、决策树模型
2D-QSAR:指纹图、分子图。可以使用一些深度学习模型。因为二维的特征其实是非常丰富的,所以支持向量机可以很好地解决高维度上的分类问题,这也是为什么用SVM调参后的结果比lightgbm要好
1D-QSAR


但是在这里我们更多的是在捕捉1D-QSAR的特征(13个),并没有很好地捕捉到2D-QSAR的特征,例如我们捕捉了氢键的数量、双键的数量、三键的数量等,但是它并不能告诉我氢键、双键、三键本身的特性以及在这个数量下能使分子有什么特性,因此就有了下面的2D-QSAR的分子指纹的特征工程。
2D-QSAR

RDKit中文教程 — RDKit 中文教程 2020.09 文档 (chenzhaoqiang.com)
AI4S往往需要有强大的背景知识才能深入地、有效地进行特征工程,上面的链接是rdkit的中文教程

在主办方提供的跑出来的结果图中可以看到,二维特征(1024个)是比一维特征更加有效的,例如xgboost的ACC在没有调参的情况下就可以达到0.6733,若精心调参是有望上0.8的
3D-QSAR
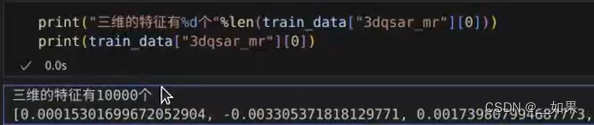
对三维的特征进行简单的拆解后可以看到一共有10000个特征,远远的大于了我们的样本量

在这种情况下我们发现其对应的ACC并不高(维度崩溃)。使用神经网络是一种比较好的改良方式
特征融合

数据方面
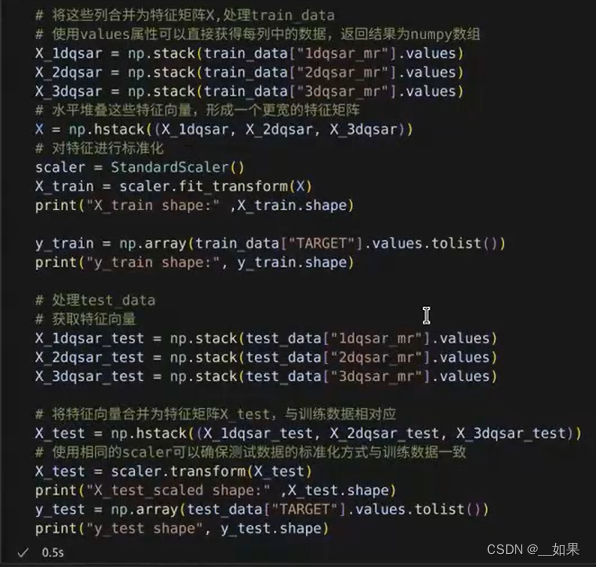
特征方面
由于特征数量与样本数量相差过大,可以考虑是否能筛选出一些重要的特征参与预测
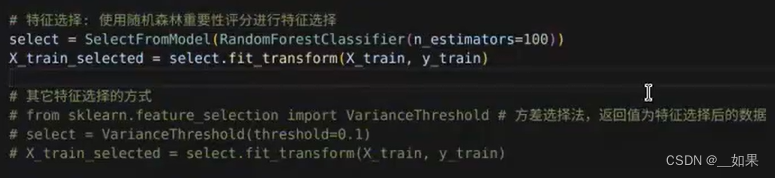
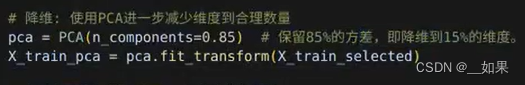
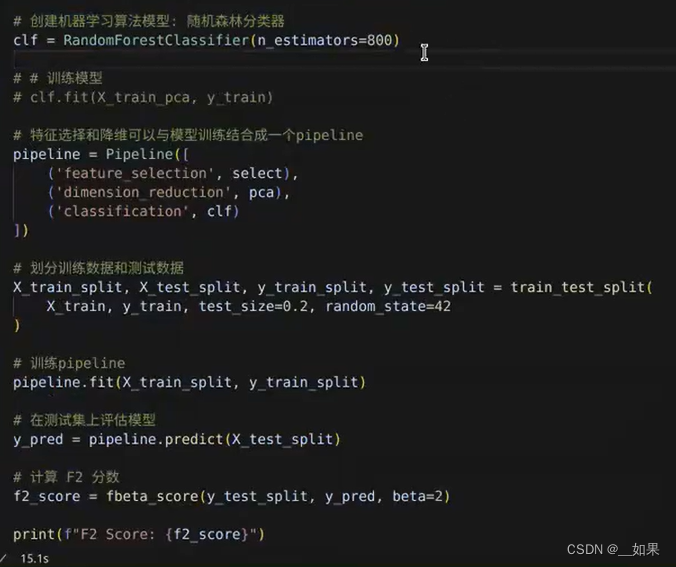
结果演示

当我们只选取两个特征:分子量与log p值时,预测结果只有0.29
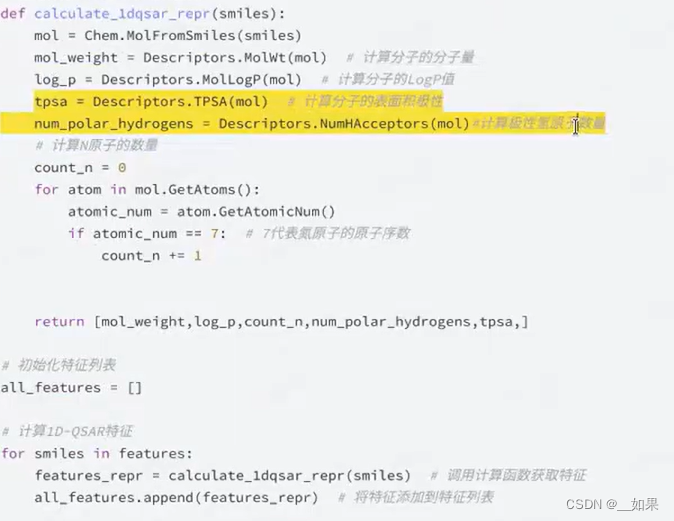
当我们选取三个特征:分子量、log p值、分子表面积极性时,预测结果有0.66分

当我们选取大量特征时,此时预测结果为0.75,并没有像之前那样大量提高分数

而我们在删去两个特征之后,发现分数反而提高了0.01。这是因为过多的特征并不会给你带来多少有效的信息,反而可能会引入不必要的噪声
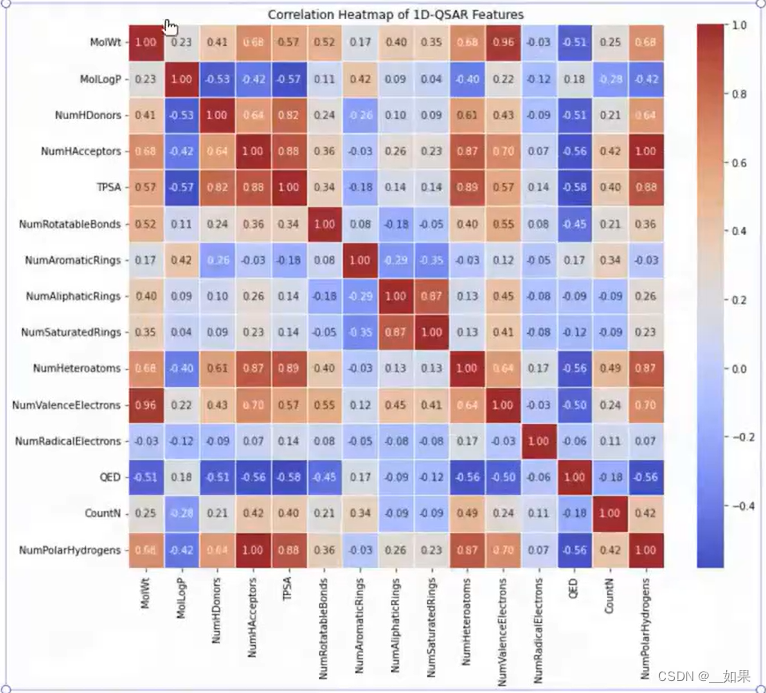
热力图分析特征之间的相关性,因为在分子领域,单独的看一种性质并不能很好地说明效果,应该更加关注不同性质的组合效果
Uni-Mol及进阶版Baseline
简介
Uni-mol是分子表示学习的框架和预训练模型,其分子表示可以自动学习,而不是像分子量、氢键数等固定的表示方法

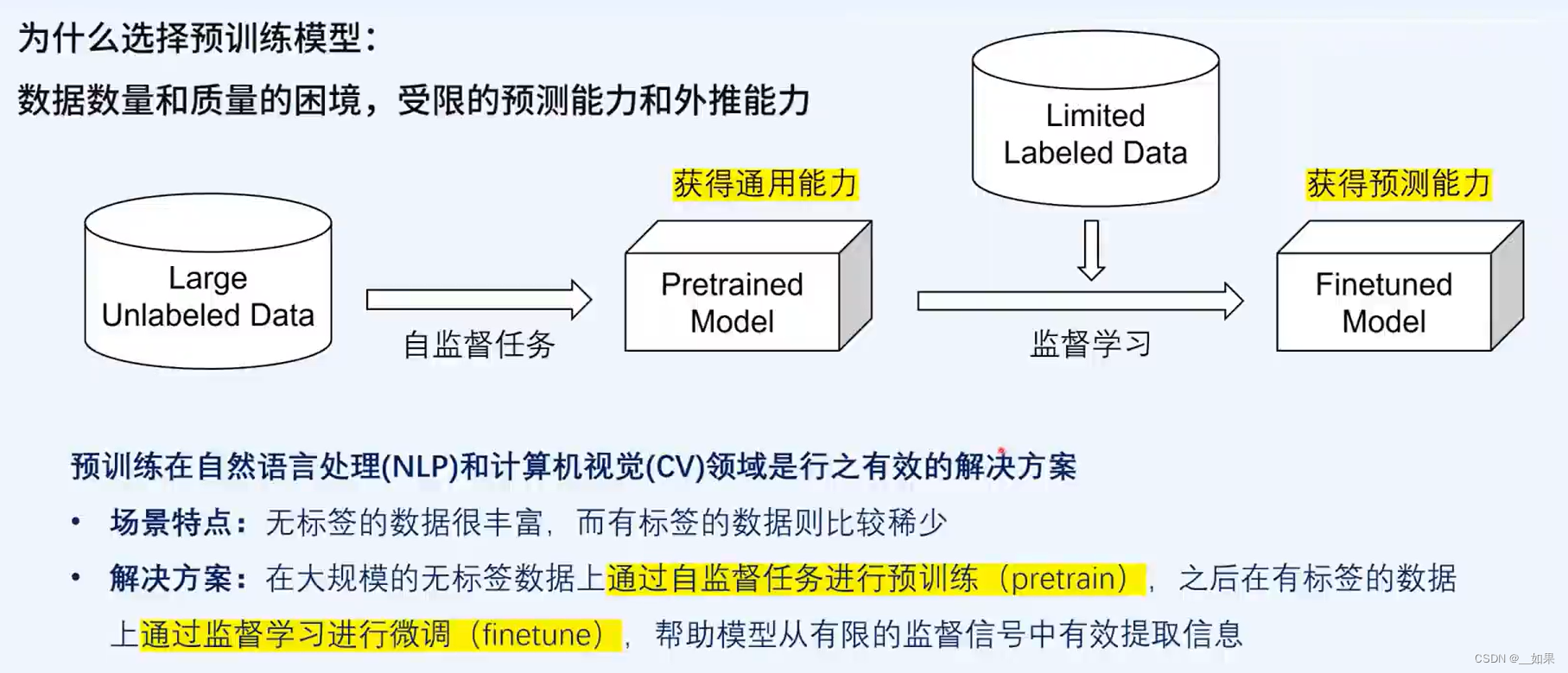
优势
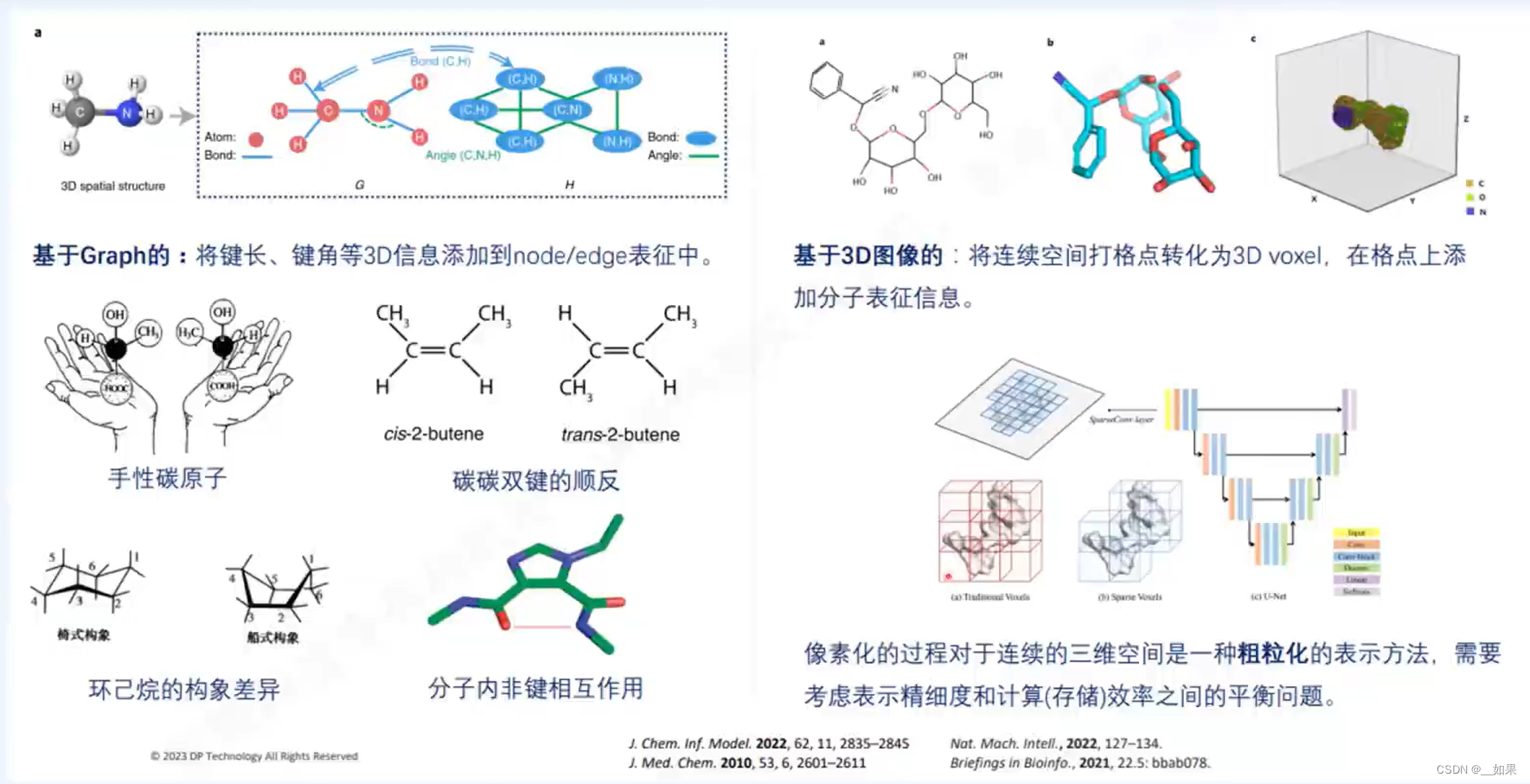
特点
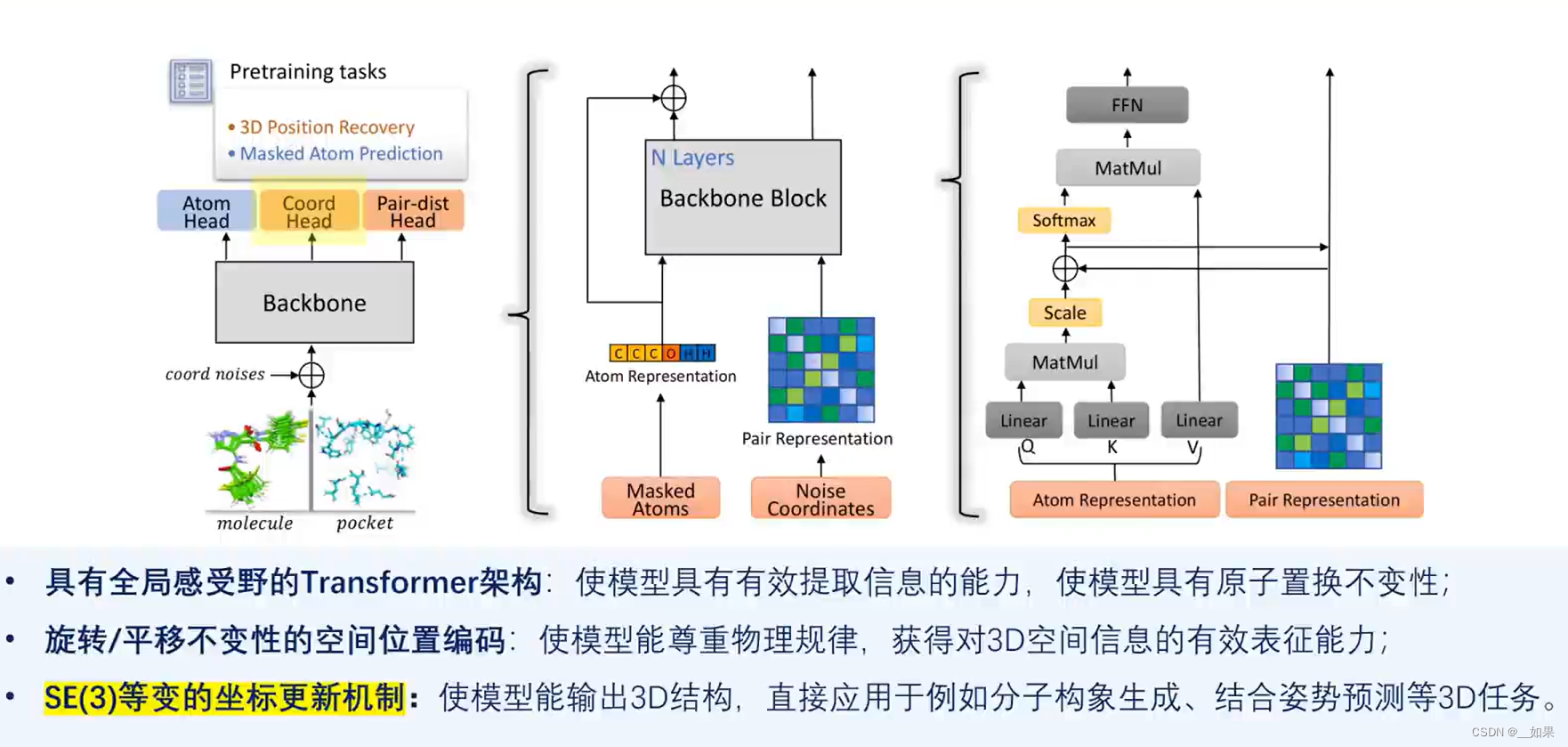

应用


进阶baseline
图神经网络
原文地址:https://blog.csdn.net/m0_73202283/article/details/134634283
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。
如若转载,请注明出处:http://www.7code.cn/show_34720.html
如若内容造成侵权/违法违规/事实不符,请联系代码007邮箱:suwngjj01@126.com进行投诉反馈,一经查实,立即删除!





![[软件工具]文档页数统计工具软件pdf统计页数word统计页数ppt统计页数图文打印店快速报价工具](https://img-blog.csdnimg.cn/direct/09dfbaff3e9a47a9a551dd65fef5d482.jpeg)