本文介绍: PackagepaleobioDBpaleobioDB 包在2020年已经停止更新,该包依赖PBDB v1 API。可以选择在下载安装包后,执行本地安装。
paleobioDB 包在2020年已经停止更新,该包依赖PBDB v1 API。
可以选择在Index of /src/contrib/Archive/paleobioDB (r-project.org)下载安装包后,执行本地安装。
Usage
Arguments
参数【data】:输入的数据,数据帧格式。可以通过 pbdb_occurrences() 函数获得数据。
参数【do.plot】:TRUE/FALSE。默认为 TRUE。
参数【col】:改变图中柱子的颜色。默认为 skyblue2。
Value
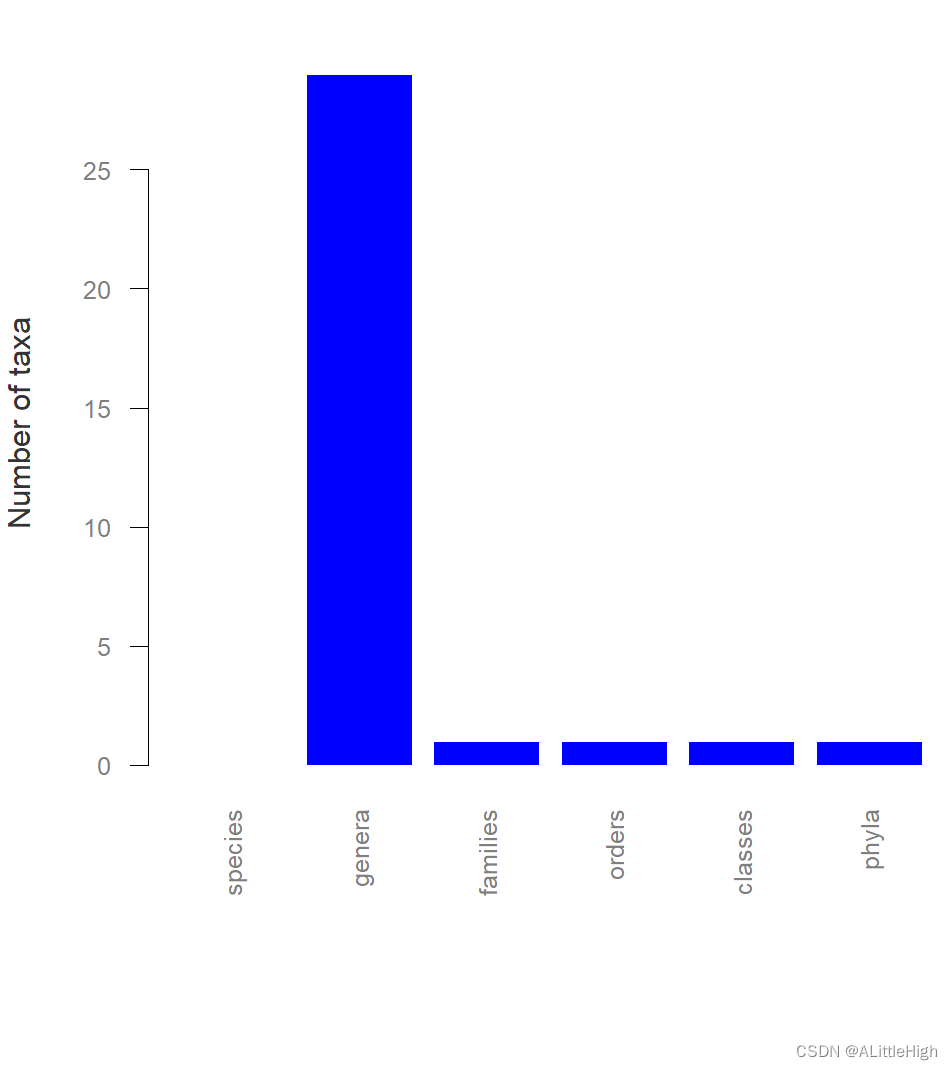
返回一张图和一个数据帧,展示了数据中的子类群数量。
Example
Page
声明:本站所有文章,如无特殊说明或标注,均为本站原创发布。任何个人或组织,在未征得本站同意时,禁止复制、盗用、采集、发布本站内容到任何网站、书籍等各类媒体平台。如若本站内容侵犯了原著者的合法权益,可联系我们进行处理。