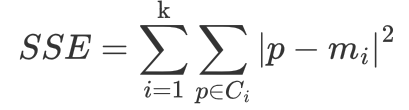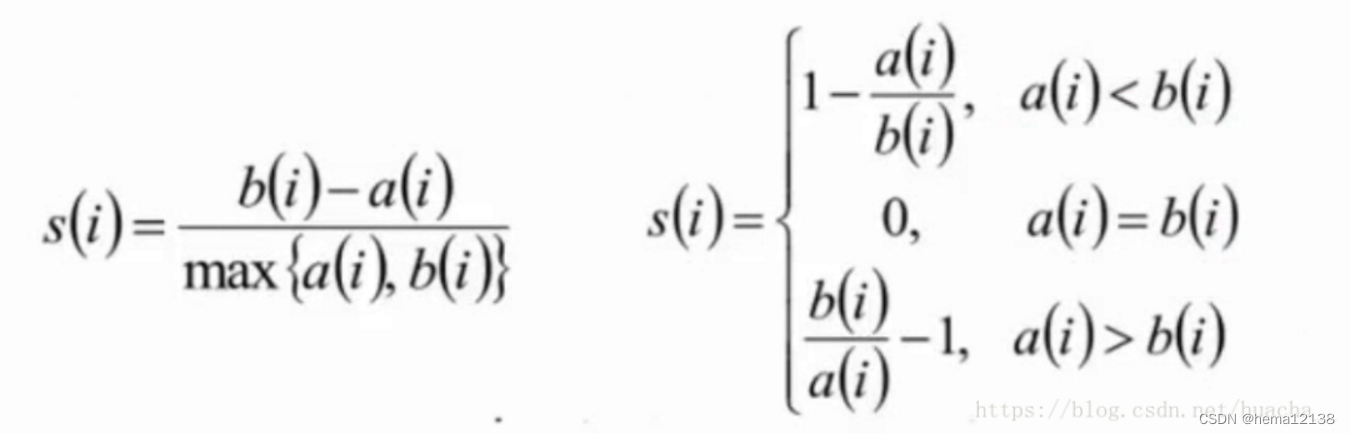其中,
i
i
i 表示向量的第
i
i
i 个分量。
我们要实现一个可以计算多个样本组成的矩阵
X
X
X,与某一个类中心
y
y
y 之间欧氏距离的函数。
给定输入矩阵
X
∈
R
n
×
m
X in mathbb{R}^{n times m}
X∈Rn×m,其中
n
n
n 是样本数,
m
m
m 是特征数,给定输入的类簇中心
y
∈
R
m
y in mathbb{R}^m
y∈Rm。
我们要计算
n
n
n 个样本到某一类簇中心
y
y
y 的欧式距离,最后的结果是
E
∈
R
n
E in mathbb{R}^{n}
E∈Rn,每个元素表示矩阵
X
X
X 中的每个样本到类中心
y
y
y 的欧式距离。
def compute_distance(X, y):
'''
计算样本矩阵X与类中心y之间的欧氏距离
Parameters
----------
X, np.ndarray, 样本矩阵 X, 维度:(n, m)
y, np.ndarray, 类中心 y,维度:(m, )
Returns
----------
distance, np.ndarray, 样本矩阵 X 每个样本到类中心 y 之间的欧式距离,维度:(n, )
'''
# YOUR CODE HERE
y= np.tile(y, (X.shape[0], 1))
distance = np.sum(((X-y)**2),axis=1)**0.5
return distance
# 测试样例
print(compute_distance(np.array([[0, 0], [0, 1]]), np.array([0, 1]))) # [ 1. 0.]
print(compute_distance(np.array([[0, 0], [0, 1]]), np.array([1, 1]))) # [ 1.41421356 1. ]
下面开始实现K-means聚类算法
class myKmeans:
def __init__(self, n_clusters, max_iter = 100):
'''
初始化,三个成员变量
Parameters
----------
n_clusters: int, 类簇的个数
max_iter, int, default 100, 最大迭代轮数,默认为100
'''
# 表示类簇的个数
self.n_clusters = n_clusters
# 表示最大迭代次数
self.max_iter = int(max_iter)
# 类簇中心
self.centroids = None
def choose_centroid(self, X):
'''
选取类簇中心
Parameters
----------
X: np.ndarray, 样本矩阵X,维度:(n, m)
Returns
----------
centroids: np.ndarray, 维度:(n_clusters, m)
'''
centroids = X[np.random.choice(np.arange(len(X)), self.n_clusters, replace = False), :]
return centroids
def compute_label(self, X):
'''
给定样本矩阵X,结合类中心矩阵self.centroids,计算样本矩阵X内每个样本属于哪个类簇
Parameters
----------
X: np.ndarray, 样本矩阵X,维度:(n, m)
Returns
----------
labels: np.ndarray, 维度:(n, )
'''
# 将每个样本到每个类簇中心的距离存储在distances中,每行表示当前样本对于不同的类中心的距离
distances = np.empty((len(X), self.n_clusters))
# 遍历类中心,对每个类中心,计算所有的样本到这个类中心的距离
for index in range(len(self.centroids)):
# 计算样本矩阵X所有样本到当前类中心的距离,存储在distances中的第index列中
# YOUR CODE HERE
distances[:, index] = compute_distance(X, self.centroids[index])
# 取distances每行最小值的下标,这个下标就是这个样本属于的类簇的标记
# YOUR CODE HERE
labels = np.argmin(distances, axis=1)
# 返回每个样本属于的类簇的标记
return labels
def fit(self, X):
'''
聚类,包含类中心初始化,类中心优化两个部分
Parameters
----------
X: np.ndarray, 样本矩阵X,维度:(n, m)
'''
# 类中心随机初始化
self.centroids = self.choose_centroid(X)
# 优化self.max_iter轮
for epoch in range(self.max_iter):
# 计算当前所有样本的属于哪个类簇
labels = self.compute_label(X)
# 重新计算每个类簇的类中心
for index in range(self.n_clusters):
# 重新计算第 index 个类中心,对属于这个类簇的样本取均值
# YOUR CODE HERE
self.centroids[index, :] = X[labels == index].sum(axis=0) / X[labels == index].shape[0]
4. 聚类
# 初始化一个3类簇的模型
model = myKmeans(3)
# 对X进行聚类,计算类中心
model.fit(X)
# 计算X的类标记
prediction = model.compute_label(X)
5. 聚类结果可视化
# 使用我们的预测结果上色
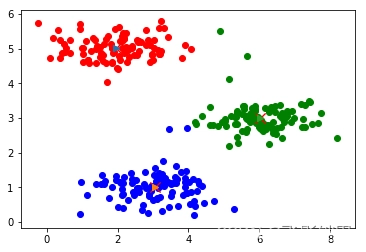
plt.scatter(X[:, 0], X[:, 1], c = prediction)
6. 评价指标
这里,我们选用两个外部指标,FMI和NMI。
from sklearn.metrics import normalized_mutual_info_score
from sklearn.metrics import fowlkes_mallows_score
print(normalized_mutual_info_score(y, prediction))
print(fowlkes_mallows_score(y, prediction))
Test
使用下面提供的数据,完成以下实验:
- 使用myKmeans和层次聚类算法(AgglomerativeClustering)对该数据进行聚类。
- 计算出两个模型的FMI和NMI值,并对聚类结果可视化。
- 分析为什么两个模型的聚类效果会出现如此的不同。
要求:
- 层次聚类的连接方式选择’single’,即使用两个类簇之间的最小距离
- 类簇个数设定为2
完成下表的填写:
双击此处填写
| 算法 | FMI | NMI |
|---|---|---|
| myKmeans | 0.4996 | 0.0003 |
| AgglomerativeClustering | 1.0000 | 1.0000 |
from sklearn.datasets import make_circles
X, y = make_circles(n_samples = 1500, factor = .5, noise = .05, random_state = 32)
plt.scatter(X[:, 0], X[:, 1], c = y)
# 初始化一个2类簇的模型
model_test = myKmeans(2)
# 对X进行聚类,计算类中心
model_test.fit(X)
# 计算X的类标记
prediction = model_test.compute_label(X)
plt.figure(figsize=(12, 4))
plt.subplot(1, 2, 1)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap='viridis', s=40)
plt.title("True Clusters")
plt.subplot(1, 2, 2)
plt.scatter(X[:, 0], X[:, 1], c=prediction, cmap='viridis', s=40)
plt.title("Agglomerative Clustering")
plt.show()
# 计算评估指标
fmi1 = fowlkes_mallows_score(y, prediction)
nmi1 = normalized_mutual_info_score(y, prediction)
print(f"K-means - Fowlkes Mallows Index: {fmi1:.4f}")
print(f"K-means - Normalized Mutual Info: {nmi1:.4f}")
# YOUR CODE HERE
agg2 = AgglomerativeClustering(n_clusters=2, linkage='single')
labels2 = agg2.fit_predict(X)
plt.figure(figsize=(12, 4))
plt.subplot(1, 2, 1)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap='viridis', s=40)
plt.title("True Clusters")
plt.subplot(1, 2, 2)
plt.scatter(X[:, 0], X[:, 1], c=labels2, cmap='viridis', s=40)
plt.title("Agglomerative Clustering")
plt.show()
# 计算评估指标
fmi2 = fowlkes_mallows_score(y, labels2)
nmi2 = normalized_mutual_info_score(y, labels2)
print(f"层次聚类 - Fowlkes Mallows Index: {nmi2:.4f}")
print(f"层次聚类 - Normalized Mutual Info: {nmi2:.4f}")
原文地址:https://blog.csdn.net/2301_76616273/article/details/136002853
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。
如若转载,请注明出处:http://www.7code.cn/show_67785.html
如若内容造成侵权/违法违规/事实不符,请联系代码007邮箱:suwngjj01@126.com进行投诉反馈,一经查实,立即删除!